生物所汪海博士与美国康奈尔大学Edward Buckler院士团队合作,开发从基因组DNA序列预测基因表达调控模式的人工神经网络模型,为实现人工智能(AI)辅助定向育种奠定了基础。该成果于3月6日在线发表在《美国科学院院刊(PNAS)》上。
从苹果的Siri到谷歌的AlphaGo,人工智能技术正在以席卷态势进入公众的视野与生活。以人工神经网络为代表的最新一代人工智能技术具有比传统机器学习技术更强大的数据挖掘能力,但是,人工智能技术在基因组学研究中尚未得到广泛的应用。其中的一个需要解决的难题就是生物中具有许多具有序列高度相似性的基因家族,在训练神经网络模型时将基因随机分配到训练集和测试集中,就会导致神经网络模型优先学习DNA序列中和基因家族或进化相关的基序,而不是真正决定基因表达调控的基序。
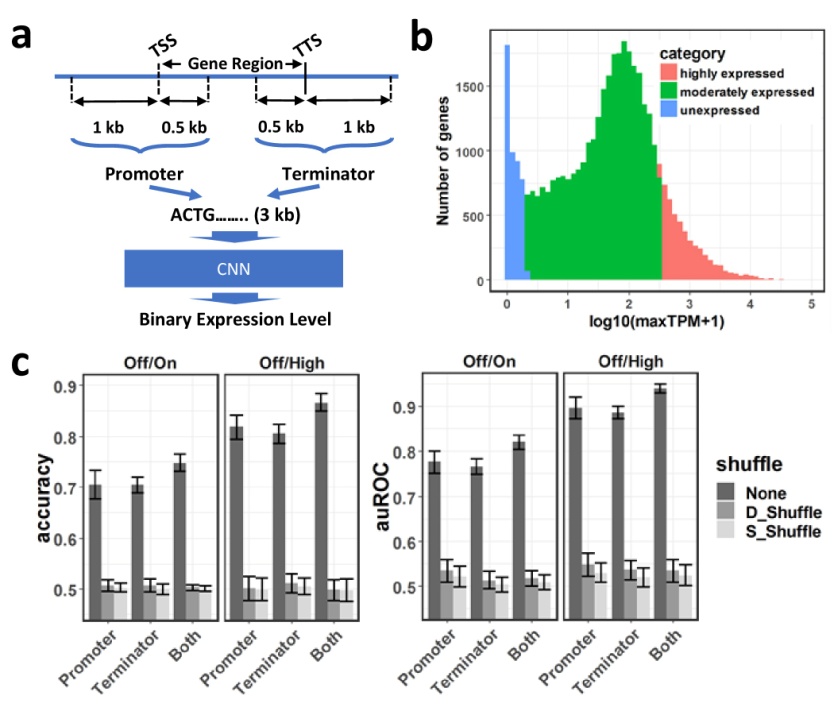
本研究中,汪海以基因家族代替单个基因为单位随机分配训练集和测试集数据,成功建立了预测二元化基因表达量(binary gene expression levels)的卷积神经网络模型。此外,进一步利用多种算法进行解析,获得了调控基因表达的关键DNA基序。在此模型的基础上,汪海的合作研究者利用进化上亲缘关系较近的两个物种,成功预测了同源基因的相对表达量,并进一步获得了调控同源基因相对表达量的关键DNA基序。
本研究建立的深度学习模型在基础理论研究和作物设计育种中具有广泛的应用前景。首先,该模型可以应用在分子生物学基础研究的各个领域,例如预测基因的时空表达特异性、转录因子结合位点、开放染色质、各种表观遗传印记、染色体重组位点等。第二,深度学习模型可以克服传统线性模型的弱点,精确预测低频/罕见变异的分子表型和田间表型效应。第三,未来可以在计算机中对基因组DNA序列进行虚拟诱变,并利用神经网络模型预测变异的后果,从中挑选符合预期目标的变异序列进行实验验证,从而实现低成本定点定向设计育种。
原文链接:https://www.pnas.org/content/early/2019/03/05/1814551116


