24日,生物所微生物蛋白设计与智造创新团队和北京畜牧兽医研究所姚斌院士团队、军事科学院军事医学研究院和河北人工智能计算中心合作,开发了首个基于华为开源全场景AI融合框架昇思(MindSpore)的蛋白质序列预训练大模型MP-BERT(基于昇思框架的蛋白双向编码器,MindSpore Protein Bidirectional Encoder Representations from Transformers),并将其应用于预测蛋白质相互作用位点,相关研究结果发表在《生物信息学简报(Briefings in Bioinformatics)》上。
本研究使用UniRef数据库中所有的蛋白质条目构造蛋白质序列(对)数据集,采用自监督方法进行模型训练,开发了一种新型蛋白质序列预训练大模型,命名为MP-BERT。这是科研人员利用华为公司研发的开源全场景AI融合框架昇思MindSpore,配合昇腾Ascend AI硬件加速运算开发的首个蛋白质序列预训练大模型,在该领域实现了硬件和软件的全国产化替代。MP-BERT作为蛋白质基座模型,只需选用合适的数据集并对下游任务进行微调,即可完成针对特定任务的模型的训练,具有广泛的应用前景。
将MP-BERT应用于预测蛋白质相互作用位点,采用预训练+微调的迁移学习策略,科研人员开发了仅基于蛋白质序列即可预测PPIs和PPI sites的方法,即用包含蛋白质及其交互点的数据集对MP-BERT进行微调,并进一步创建了MPB-PPI和MPB-PPISP两种模型,用于预测蛋白质相互作用和相互作用位点。相比于传统的实验方法和其他人工智能算法,具有成本低、效率高和准确率高的优点。
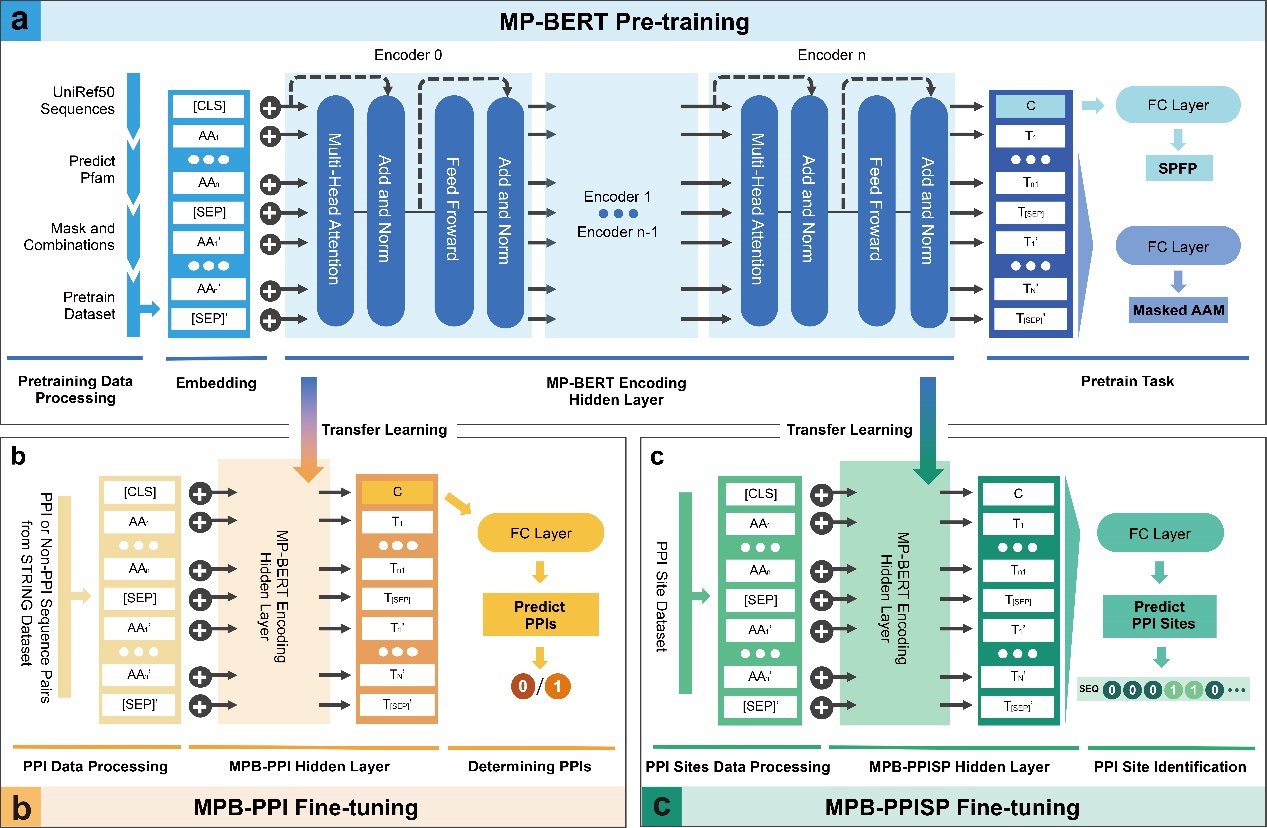
图注:MP-BERT及其微调模型架构
中国农业科学院生物技术研究所硕士生刘拓宇为论文第一作者,生物所关菲菲副研究员,军事科学院军事医学研究院滕越研究员、中国农业科学院北京畜牧兽医研究所黄火清研究员和田健研究员为论文共同通讯作者。该研究得到国家重点研发计划、国家自然科学基金和基本科研业务费专项等基金的资助。
论文链接:https://academic.oup.com/bib/article/24/6/bbad376/7326134


